 Les virus sont des parasites cellulaires obligatoires. Cela signifie qu’ils ne se multiplient qu’à l’intérieur d’une cellule. La cellule procure toutes les fonctions (enzymes), tous les ingrédients (acides aminés, bases, lipides etc.), ainsi que l’énergie nécessaire pour produire les composants des nouvelles particules virales, composants dictés par l’information génétique virale. Pour faire simple, les composants viraux sont de deux sortes. Les composants dit structuraux : le génome (ADN ou ARN), plus les protéines qui composent la particule virale et les composants non structuraux qui varient selon les familles de virus. Ce sont des fonctions absentes de la cellule, comme une enzyme qui copie de l’ARN en ADN ou en ARN (ApAd, cf. ci-dessous), des fonctions permettant l’appropriation du milieu cellulaire par le virus, ou des fonctions qui contrecarrent les mécanismes de défense de la cellule et de l’organisme infectés. Dans cette confrontation virus-cellule, le virus évolue vers une multiplication optimale dans l’environnement cellulaire à disposition. Ne pouvant pas influencer cet environnement, le virus s’y adapte au mieux. Le fondement de cette adaptation repose sur l’introduction de mutations qui résultent en une population virale composée en permanence d’une multitude de variants, parmi lesquels, le variant le mieux adapté à l’environnement est sélectionné , et forme la majorité de la population virale, ceci à un moment donné et dans un environnement donné (cf. La quasi-espèce).
Les virus sont des parasites cellulaires obligatoires. Cela signifie qu’ils ne se multiplient qu’à l’intérieur d’une cellule. La cellule procure toutes les fonctions (enzymes), tous les ingrédients (acides aminés, bases, lipides etc.), ainsi que l’énergie nécessaire pour produire les composants des nouvelles particules virales, composants dictés par l’information génétique virale. Pour faire simple, les composants viraux sont de deux sortes. Les composants dit structuraux : le génome (ADN ou ARN), plus les protéines qui composent la particule virale et les composants non structuraux qui varient selon les familles de virus. Ce sont des fonctions absentes de la cellule, comme une enzyme qui copie de l’ARN en ADN ou en ARN (ApAd, cf. ci-dessous), des fonctions permettant l’appropriation du milieu cellulaire par le virus, ou des fonctions qui contrecarrent les mécanismes de défense de la cellule et de l’organisme infectés. Dans cette confrontation virus-cellule, le virus évolue vers une multiplication optimale dans l’environnement cellulaire à disposition. Ne pouvant pas influencer cet environnement, le virus s’y adapte au mieux. Le fondement de cette adaptation repose sur l’introduction de mutations qui résultent en une population virale composée en permanence d’une multitude de variants, parmi lesquels, le variant le mieux adapté à l’environnement est sélectionné , et forme la majorité de la population virale, ceci à un moment donné et dans un environnement donné (cf. La quasi-espèce).
Ce préambule terminé et fort de la compréhension du code génétique (cf. Le code génétique), voyons dans plus de détails le bal des mutations et de leurs effets, en prenant, au hasard, l’exemple du SARS-CoV-2. Le génome du SARS-CoV-2 est une molécule d’ARN comprenant une séquence de 29’903 nucléotides (nt, symbolisées par les lettres U, C, A, G). Lors de la copie de ce génome, au cours de la multiplication du virus, cette séquence est d’abord copiée en son complément où U est copié en A, C en G, A en U et G en C, donnant naissance à une séquence intermédiaire, complémentaire du génome. Cet intermédiaire va être copié de multiples fois en son complément (avec le même appariement: U-A, C-G, A-U, G-C). Ces copies représentent autant de nouveaux génomes (on parle de réplication du génome). C’est durant cette opération de réplication que les mutations sont introduites.
Les mutations les plus fréquentes, dites ponctuelles, résultent en une erreur d’appariement lors de la copie.
La vidéo 1 illustre une copie conforme d’une minuscule partie du génome. La copie du génome (noir) par l’ARN polymérase ARN dépendante (ApAd, cf. Produire de médicaments antiviraux) résulte en un intermédiaire (vert) dont la séquence est le complément du génome selon le bon appariement (cf. ci-dessus). Cette séquence intermédiaire est ensuite copiée selon le même principe en son complément (bleu), qui représente la copie conforme du génome (comparez bleu et noir).
La vidéo 2 montre la même opération avec cette fois une erreur de copie. Le A, 11ème lettre de la séquence présentée, au lieu d’être copié en U est faussement copié en C dans la séquence intermédiaire. Ce C est ensuite copié en G dans l’étape suivante, générant un génome modifié où la séquence initiale diffère d’une lettre de la séquence copiée. C’est l’introduction d’une mutation ponctuelle. Cette erreur est programmée pour se produire de manière aléatoire (à n’importe quelle position de la séquence) en moyenne environ 1 fois tous les 10’000 lettres copiées. Cette fréquence de mutations correspond, pour des génomes à ARN dont la longueur moyenne est de 10’000 lettres, à l’introduction d’une mutation par événement de réplication. Ainsi, il est probable qu’il n’y ait pas production de deux génomes absolument identiques lors de la multiplication virale dans un organisme (*voir la note en fin d’article pour la particularité des Coronavirus).
Mais alors, vous direz-vous, comment est-ce possible qu’il y ait un virus avec une identité précise qui se propage. Cela tient au fait que tous ces variants génétiques sont en permanence sélectionnés par l’environnement cellulaire pour faire ressortir le génome le mieux adapté. Et pour un environnement précis, il y en a souvent un seul, le mieux adapté, celui qui constitue la majorité des génomes. Tous les autres, soit végètent au niveau où ils sont produits (1/10’000), soit finissent par disparaître parce qu’ils ne sont pas viables. Pourtant, c’est sur la population de variants viables minoritaires que repose le pouvoir d’adaptation. Au cas où il y a changement d’environnement cellulaire, la solution est présente dans la population de variants, qui pour être minoritaire, n’en est pas moins extrêmement variée.
La vidéo 3 présente les conséquences de l’introduction d’une mutation ponctuelle. Comme vu précédemment (cf. Information génétique…), la séquence de lettres prend son sens lorsqu’elle est lue en mots de trois lettres, les codons, auxquels correspondent des acides aminés, les constituants des protéines. En 3a. la séquence de 18 lettres est groupée en six mots de trois lettres. Ces six codons sont lus par la machinerie cellulaire (rib, pour ribosome) qui les interprète et produit la séquence correspondante d’une partie de protéine comprenant les acides aminés indiqués, ceci selon le code génétique: CUU –>leucine (Leu), UAU –> tyrosine (Tyr) etc. En 3b., une mutation ponctuelle est intervenue sur la troisième lettre du deuxième codon qui a changé de U à C. Cette mutation n’a pas de conséquence car les deux codons UAU et UAC sont décodés en un acide aminé identique, la tyrosine (Tyr). On dit que la mutation est synonyme, elle a remplacé un mot (codon) par un autre de signification identique, grâce à la redondance du code génétique. En 3c., la même lettre de deuxième codon a été changée en A, générant le codon UAA, qui signifie arrêt de lecture. La synthèse de la protéine s’arrête à ce codon Stop : toute la partie de la protéine en aval va manquer. La fonction de cette protéine affiche une très haute probabilité d’être annulée par cette mutation. La mutation ponctuelle va ici priver le virus d’une fonction, qui aura un effet négatif marqué sur la multiplication virale, le virus ne sera probablement pas viable. Finalement en 3d., la mutation ponctuelle s’est produite dans le 4ème codon où GAU a été transformé en GGU. De ce fait, l’acide aspartique (Asp) a été remplacé par la glycine (Gly). C’est de loin le cas le plus intéressant, car la protéine est produite sous une forme modifiée. La fonction peut être conservée et cette modification peut représenter, le cas échéant, une solution vers une meilleure adaptation.
La courte séquence présentée ci-dessus est empruntée au génome du SARS-CoV-2, et plus précisément dans la partie du génome qui dirige la production de la protéine S, le spicule à la surface de la particule virale.
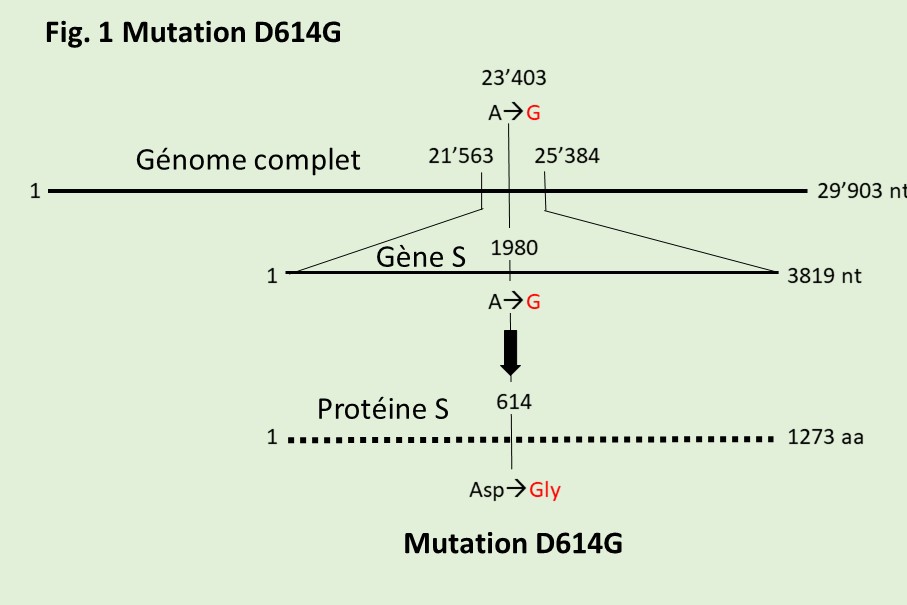
La Fig. 1, situe plus précisément ce gène S qui s’étend des lettres 21’563 à 25’384, dans le génome de 29’903 lettres (nt= nucléotides=bases=lettres). Dans ce gène de 3819 lettres, en position 1980, la mutation de A en G a eu lieu. La répercussion de cette mutation sur la composition de la protéine est montrée en dessous. Dans la protéine, longue de 1273 acides aminés (aa), l’acide aminé acide aspartique, en position 614, (Asp) est changé en glycine (Gly). Comme l’abréviation de Asp est D, et celle de Gly est G, on parle de la mutation D614G. Et si on en parle, c’est parce que dans la vraie vie cette mutation s’est produite et que le variant G614 est en train de se propager, parfois aux dépens du virus originel, D614.
Avant de parler des effets de cette mutation, il vaut la peine de s’arrêter sur le processus qui a conduit ce variant minoritaire à émerger dans la population virale. On sait maintenant (cf. ci-dessus) que cette mutation est le résultat d’une erreur d’appariement aléatoire au cours de la multiplication du génome viral. Une fois produit, le virus G614 a passé par l’étape de sélection, à l’œuvre en permanence. Selon toute évidence, cette étape a mis en évidence une propriété qui donne à G614 un avantage (on parle d’avantage sélectif). Ce peut-être une production de virus plus élevée, une propagation de cellules à cellules plus efficace, une stabilité de la particule virale plus grande, un pouvoir infectieux entre individus plus soutenu etc. Plus, plus, plus…. plus que quoi ? Plus que le virus qui constituait l’espèce majoritaire (D614) jusqu’à son apparition ou du moins tout autant. Le reste est affaire d’épidémiologie. On peut distinguer au moins deux situations. Soit G614, une fois produit dans un individu, a été transporté dans un lieu géographique nouveau et s’y est répandu efficacement, on parle d’un événement fondateur. Une île, un pays, une région aura donc « son » virus qui diffère du virus originel, D614, présent en d’autres lieux. Soit G614 émerge dans une population où D614 circule. Les deux virus sont en compétition et si G614 est mieux adapté que D614, il va prendre sa place, à moins que les deux virus ne cohabitent. Le profil épidémiologique montre les deux cas de figure.
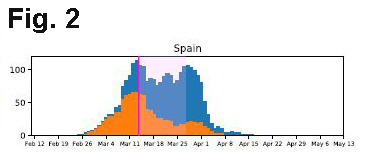
La Fig. 2. illustre le cas où, dans le moment de l’observation, les deux virus cohabitent dans une région d’Espagne, bien qu’à des niveaux différents. Difficile à dire qui a précédé qui. Le G614 (en bleu) semble se propager avec plus d’efficacité.
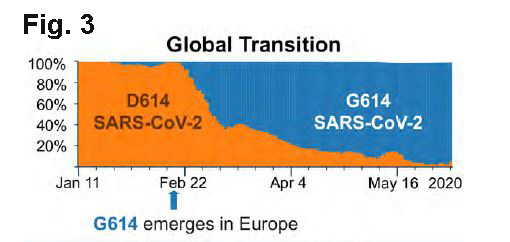
La Fig. 3. montre au contraire un exemple où le G614 prend clairement avec le temps la place du D614. Ceci à l’échelle d’un continent. Le D614 (en orange), qui constitue le 100% des cas analysés au début de la pandémie, disparaît en trois mois au profit du G614.
La Fig. 4. Illustre encore une autre situation où le G614 semble avoir été introduit dans diverses régions d’Italie et de France par des événements fondateurs qui se reproduisent en différents lieux en d’autres temps.
Ces profils ont valeur d’exemples. Ils sont indicateurs de différences de propagation, mais ne prétendent pas les démontrer. D’ailleurs les nombres de cas répertoriés sont pour la plupart trop faibles pour avoir valeur démonstrative.
G614 a été démontré, en laboratoire, produire plus de particules virales que D614. Il ne semble pas présenter une carte d’identité différente face à des anticorps neutralisant, ce qui est une bonne nouvelle dans l’optique de la mise au point d’un vaccin. Il ne semble pas associé avec des symptômes différents (plus graves, moins graves) que le virus D614. L’histoire est en plein développement et les caractéristiques proposées ici et maintenant demandent à être confirmées ou revues à intervalle régulier. En guise de conclusion, la mutation D614G apparaît également (de plus en plus ?) associée à une panoplie de 4-5 autres mutations, ce qui n’est pas pour simplifier l’appréhension de la propagation du SARS-Cov-2 et de ses caractéristiques.
D614G est une mutation qui n’émerge pas comme une solution à un changement de l’environnement, mais résulte plutôt d’une sélection d’un variant plus à même de coloniser l’environnement à disposition. L’émergence de SARS-CoV-2 dans la population humaine a impliqué une première adaptation, qui peut être décrite par comparaison avec le virus animal (chauve-souris) dont il est issu. Ensuite, dans ce nouvel environnement, il y a de la place pour « affiner » l’adaptation. C’est probablement dans ce cadre qu’il faut placer la mutation D614G.
En l’absence de médicaments et de vaccins, on pourrait imaginer que les stratégies de lutte adoptées pour limiter la propagation du virus orientent l’adaptation. En effet, chaque fois qu’un individu gravement infecté est isolé et guérit (ou décède) de son infection, la population virale qui l’infecte disparaît de la scène. En revanche, les porteurs asymptomatiques continuent à propager le virus. Dans le lot de virus qu’ils véhiculent, y a-t-il des souches virales moins pathogènes ? Si c’était le cas, elles viendraient peu à peu remplacer les souches plus virulentes. C’est en fait une notion acceptée en virologie que l’évolution de la relation virus-hôte tende vers une coexistence pacifique. Pour l’instant, hélas, des souches virales du SARS-CoV-2 affichant une virulence moindre dans le cas des infections asymptomatiques n’ont pas été mises en évidence.
Les étapes d’utilisation de médicaments spécifiques et de vaccination vont représenter, lorsque/si elles se matérialisent, des changements d’environnement drastiques pour le virus. Il est quasiment certain que des résistances au médicament vont émerger. Quant à la stabilité de la carte d’identité du virus, la question reste ouverte. SARS-CoV-2 va-t-il se comporter comme les virus de la rougeole ou de la poliomyélite, pour lesquels les vaccins préparés dans les années 1950-60 sont toujours d’actualité ou comme les virus de la grippe, pour lesquels une adaptation des souches vaccinales doit se faire à intervalles réguliers. Affaire à suivre.
*Nota bene : Il avait été pressenti que des génomes de virus à ARN plus longs que 15’000 à 20’000 lettres ne pourraient pas exister, car cela impliquerait un nombre de mutations par génome répliqué trop élevé (>2). Les Coronavirus, avec des génomes de 30’000 à 35’000 lettres ont démenti cette prévision. C’est que ces virus ont développé un système de correction des erreurs de l’AdAp, qui ramène le taux de mutation dans l’ordre de celui des virus à ARN au génome plus court, qui eux n’ont pas besoin de ce système, leur évolution ayant réglé au juste niveau leur taux de mutations.