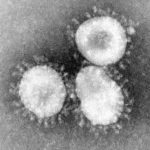
 Les Coronavirus tiennent leur nom de l’aspect de couronne que prend la particule virale vue au microscope électronique. Ils appartiennent à l’ordre des Nidovirales, famille des Coronaviridae, sous-famille des Coronavirinae. Cette sous famille est encore divisée en quatre genres les alpha-, beta-, gamma- et delta-coronavirus. Cette classification est fondée sur des particularités biologiques comprenant le mode de transmission, la stratégie d’infection, la stratégie de multiplication et/ou d’interactions avec l’hôte infecté etc. Cette classification a comme but de poser un contexte où l’humain n’est qu’un des hôtes (occasionnel ou pas) d’une très large population de virus.
Les Coronavirus tiennent leur nom de l’aspect de couronne que prend la particule virale vue au microscope électronique. Ils appartiennent à l’ordre des Nidovirales, famille des Coronaviridae, sous-famille des Coronavirinae. Cette sous famille est encore divisée en quatre genres les alpha-, beta-, gamma- et delta-coronavirus. Cette classification est fondée sur des particularités biologiques comprenant le mode de transmission, la stratégie d’infection, la stratégie de multiplication et/ou d’interactions avec l’hôte infecté etc. Cette classification a comme but de poser un contexte où l’humain n’est qu’un des hôtes (occasionnel ou pas) d’une très large population de virus.
- Les alphacoronavirus ont été identifiés chez les chats, les chiens, les porcs, et l’humain (HCoV-229E).
- Les betacoronavirus ont été trouvés occasionnellement dans le bétail, les souris et les rats, les porcs, l’humain (HCoV-HKU1, HCoV-OC43, HCo-V-NL63) et les chauves-souris. C’est dans ce genre que sont classés le SRAS, et le MERS, les deux virus qui ont émergé dans la population humaine en 2003 et 2012, respectivement (cf. Emergence d’un virus :Saison III…). C’est dans ce genre qu’est classé le SARS-CoV-2 (en français SRAS-CoV-2) qui sévit en ce moment, dont la maladie qu’il provoque a été nommée COVID-19.
- Les gammacoronavirus ont été identifiés principalement dans le monde aviaire, ainsi que
- les deltacoronavirus qui eux infectent également les mammifères.
A l’exception des SARS-CoV, SARS-CoV-2 et MERS-CoV, les coronavirus humains ont régulièrement causé un faible pourcentage d’infections respiratoires plutôt bénignes chaque année. Ils peuvent également atteindre le foie, l’intestin, voire le système neurologique.
Les Coronavirus sont des virus à ARN. Leur génome n’est ainsi pas composé de molécules d’ADN, comme le sont les génomes de tous les organismes vivants, unicellulaires (bactéries, levures..) ou pluricellulaires (humain, ver de terre…), comme le sont les génomes d’une multitude d’autres virus, dits virus à ADN (virus herpétiques, adénovirus ou papillomavirus (cf. Le génome). Parmi les virus à ARN humains les plus connus on trouve le VIH, les virus de la grippe, de la rougeole, de la poliomyélite, du rhume etc…De manière intéressante, tous les virus qui ont affiché la capacité d’émerger dans la population humaine, depuis que nous sommes en mesure de les identifier, ont été et restent des virus à ARN. La nature du génome ne semble donc pas étrangère à la possibilité d’émergence. Ceci peut s’expliquer par la capacité d’adaptation qu’il faut afficher pour changer d’espèce d’hôtes. Le contexte biologique humain n’est pas identique à celui de la chauve-souris, du chat ou même du singe. Pour un virus, franchir la barrière d’espèce signifie survivre dans un monde inconnu. Cela nécessite une capacité à s’adapter à des caractéristiques biologiques nouvelles. Cette capacité vient de la plasticité génétique que confère la multiplication d’un génome composé d’ARN.
La multiplication virale implique l’infection d’une cellule et la production de masse de tous les composants viraux qui vont s’assembler pour produire de nouvelles particules virales (cf. Un virus, ça se multiplie…). Un virus entre dans la cellule, des milliers de virus en sortent, le génome infectant doit être multiplié autant de fois. Le génome est ainsi recopié de multiples fois, ces génomes recopiés sont eux-mêmes recopiés le nombre de fois suffisant pour atteindre ces milliers de copies qui vont faire partie des milliers de nouvelles particules virales. Il se trouve que la copie d’ARN pour produire un nouvel ARN n’est pas une fonction cellulaire. La cellule peut copier l’ADN pour produire de l’ARN (et de l’ADN…), mais ne possède pas de machinerie pour copier l’ARN en ARN. Cette fonction, c’est le virus qui doit l’amener dans son bagage. Autrement dit, le génome viral doit contenir l’information pour produire cette machinerie, une ARN polymérase qui copie l’ARN en ARN, une ARN polymérase ARN dépendante (ApAd). Il se trouve que cette ApAd virale a évolué pour ne pas faire des copies parfaites, mais pour introduire de manière aléatoire des erreurs. La vidéo 1 illustre le travail de cette machinerie.
Le génome est symbolisé par la séquence d’une dizaine de bases (en noir). L’ApAd doit d’abord faire une copie intermédiaire du génome (en vert). Pour le faire l’ApAd, copie un A en U, un U en A, un C en G et un G en C. Cet intermédiaire est recopié dans un deuxième temps pour produire une copie du génome (en bleu). La vidéo 2 illustre l’imprécision de la ApAd qui, soit dans la copie du génome, soit dans la copie de l’intermédiaire, introduit une variation.
Cette variabilité a été estimée se produire à une fréquence de 1/10’000. Autrement dit chaque fois que la ApAd recopie 10’000 bases, elle a la probabilité d’introduire une variation. Sachant que les génomes des virus à ARN contiennent en moyenne 10’000 bases, la probabilité existe que chaque génome recopié contienne une variation par rapport au génome dont il est la copie. Les particules virales produites constituent donc une réserve énorme de génome variants, une population hétérogène appelée « quasi-espèce »(cf. La Quasi-espèce, une hétérogénéité..). Ces variants sont le gage d’une adaptation possible au changements de caractéristiques biologiques, comme le constitue une nouvelle espèce d’hôtes. En effet, dans la masse des virus, il va s’en trouver un dont les variations génétiques permettent une adaptation aux conditions nouvelles. Il sera sélectionné pour devenir le génome de la nouvelle population de virus. Cette sélection pour les virus le mieux adaptés se poursuit en permanence. Tous les variants qui n’apportent pas d’avantages à la population virale végètent à la fréquence à laquelle ils ont été produits (1/10’000) ou alors disparaissent.
Les Coronavirus possède un génome entre 25’000-32’000 bases. Selon les estimations, il avait été conclu qu’une telle longueur de génome à ARN ne serait pas viable, sachant que l’accumulation de plusieurs variations (mutations > 3) dans un même génome, aboutit généralement à un génome non viable, c’est à dire à un génome qui produit des virus non viables. Il se trouve que l’ApAd des coronavirus est assistée par une fonction correctrice qui vient limiter les variations (mutations) excédentaires, tout en permettant une variabilité nécessaire au changement d’espèce d’hôtes. Les coronavirus ont un deuxième mécanisme pour limiter l’accumulation contreproductive de trop de variations.
Ils sont passés maîtres dans le recyclage de deux génomes non viables (parce que contenant 4 mutations, X), en un génome viable (ne contenant que 2 mutations), par recombinaison. Cela semble une réussite si l’on se fonde sur la capacité d’émergence de ces virus.
Il semblerait même que la recombinaison ait joué un rôle majeur dans la génération du SARS-CoV-2, qui serait le produit recombiné du SARS-CoV-1 (2003) et d’un coronavirus de chauve-souris. Pour cela, il aurait fallu que les deux virus infectent à un moment donné les même cellules de l’hôte infecté.
(cf. également Émergence d’un virus: saison I et saison II).
La pandémie est déclarée. Il y a peu de chance pour que la Suisse soit épargnée. En attendant les directives de l’OFSP, on peu déjà consulter un site de Questions / Réponses qui apportent des informations utiles.