Le test RT-PCR appliqué au SARS-CoV-2 permet de détecter le génome viral présent aussi bien dans les particules virales que dans les cellules infectées par le virus.
Le test RT-PCR appliqué au SARS-CoV-2 permet de détecter le génome viral présent aussi bien dans les particules virales que dans les cellules infectées par le virus.
Commençons par dévoiler les acronymes. RT vaut pour « Reverse Transcirptase ». C’est de l’anglais. En français cela se dit « transcriptase réverse ou inverse ». PCR vaut pour « Polymerase Chain Reaction», en français, « réaction de polymérase en chaine ». RT et PCR définissent ainsi deux activités enzymatiques (une enzyme est une protéine qui effectue une réaction biochimique).
- Prenons d’abord la réaction de transcriptase réverse. En biologie moléculaire, la transcription désigne l’action de copie de l’ADN en ARN. L’information génétique contenue dans l’ADN est décryptée lorsqu’elle est transcrite en ARN par une transcriptase (aussi appelée ARN polymérase). Le flux normal de l’information génétique va ainsi d’ADN à ARN (cf. Le Génome et le Vaccin ARNm). C’est un dogme en biologie, aussi fondamental que la transsubstantiation pour les catholiques romains. Pourtant en 1970, Howard Temin et David Baltimore découvrent une transcriptase qui fait l’inverse : elle copie l’information contenue dans l’ARN en ADN. La découverte de cette activité, qui bouleverse le dogme, a valu aux deux chercheurs le Prix Nobel en Physiologie et Médecine en 1975. Cette activité est présente dans les rétro-virus (par exemple HIV) dont le génome est composé d’ARN.
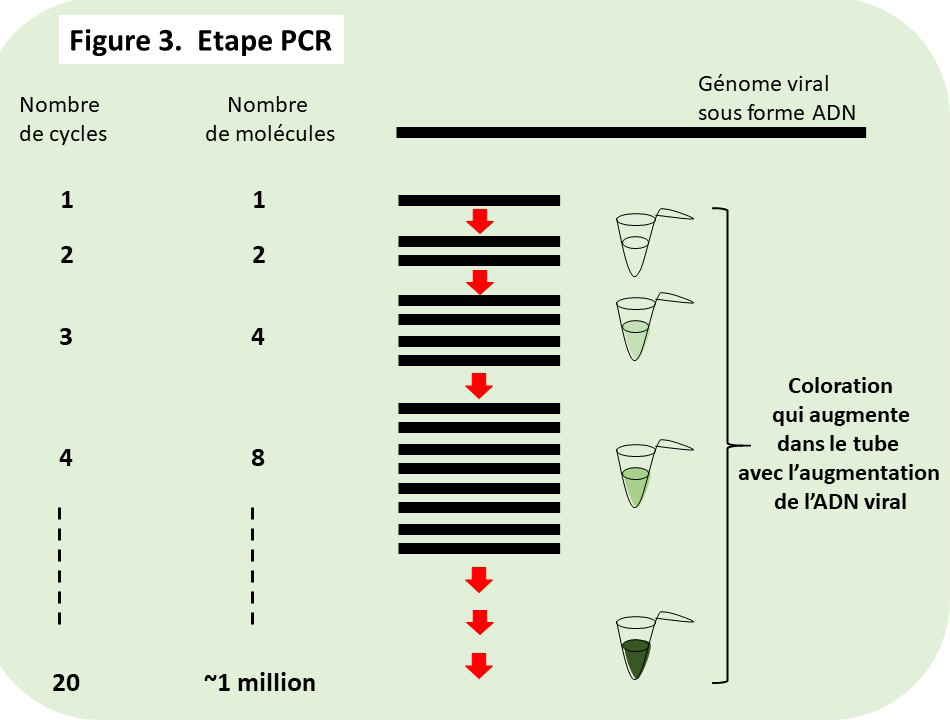
- Venons-en à la réaction de polymérase en chaine. Cette réaction implique une copie d’ADN en ADN, ceci de façon répétée, avec comme résultat une augmentation à chaque cycle de deux fois la quantité d’ADN : 1 copie donne deux copies, qui donnent 4 copies, qui donnent 8 copies etc. etc. Après 20 cycles de copie, la quantité originale d’ADN a été multipliée par près d’un million de fois. Plusieurs points sont à relever à propos de cette amplification. D’abord, cette réaction n’est possible que pour l’ADN. Elle nécessite des conditions (notamment de température élevée, de 40°C, 60C° voire 90°C) qui dégraderaient l’ARN. Ensuite cette amplification permet de faire ressortir de l’ADN présent à l’origine en très faible quantité. Finalement, cette amplification peut être rendue très sélective : est amplifié que l’ADN que l’on choisit d’amplifier (cf. ci-dessous).
Dans le test RT-PCR qui nous préoccupe ici, les deux réactions sont combinées : une réaction de RT précède la PCR. Pourquoi cette combinaison? Parce que le génome du SARS-CoV-2 est composé d’ARN. Comme tel, il n’est pas amplifiable. Il doit donc, dans un premier temps être transformé en ADN.
Ces préliminaires énoncés, passons à une description plus détaillée du test (cf. Figures 1-5).
- La Figure 1 montre le prélèvement de l’échantillon à l’aide de l’écouvillon qui vient chatouiller le fonds des fosses nasales. L’écouvillon est plongé dans un liquide biologique où, s’il y a infection, on s’attend à trouver des particules virales produites par l’infection ainsi que des cellules infectées, bourrées de génomes viraux et d’ARN cellulaires. L’ARN viral et les ARN cellulaires sont ensuite purifiés et concentrés, prêts pour l’étape suivante.
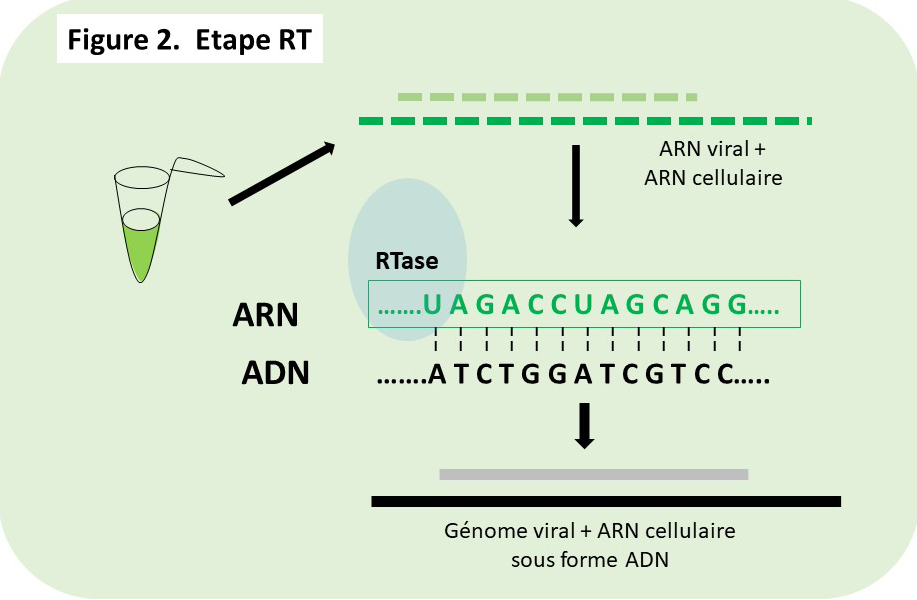
- La Figure 2 et son animation présentent la réaction de la transcriptase réverse (RTase) copiant ces ARN, indistinctement viraux ou cellulaires, et produisant les ADN correspondants.
- La Figure 3 illustre l’action de l’ADN polymérase qui duplique en série, cette fois uniquement un fragment bien précis de l’ADN viral (cf. Figure 4). A chaque réaction, le produit de polymérisation double. La production d’ADN est évalué par l’augmentation d’un colorant (visible en lumière UV) qui résulte de la consommation des réactifs nécessaires à l’activité de l’ADN polymérase (cf. Figure 5).
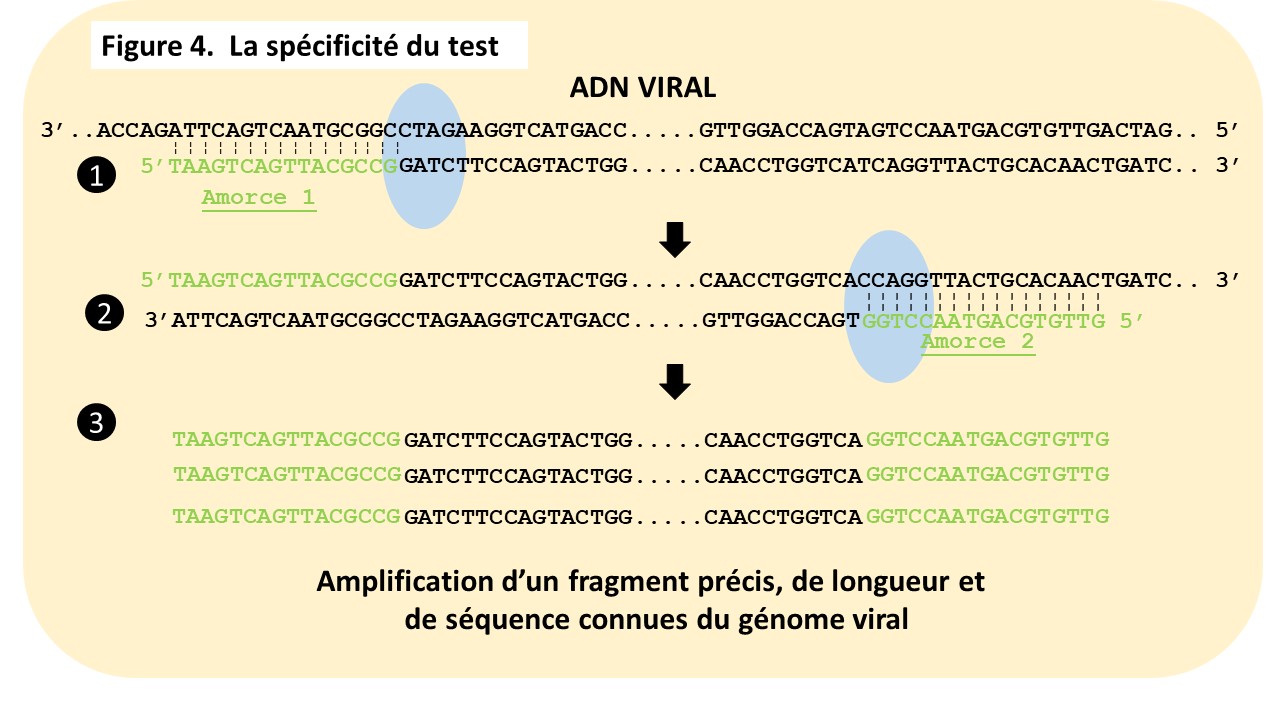
- Comment faire en sorte d’ignorer l’ADN cellulaire présent dans l’échantillon produit par la RTase (cf. Figure 2), et donner de la spécificité à cette amplification. L’explication se trouve dans la Figure 4 et son animation.
- L’ADN polymérase (en bleu), pour commencer son travail de copie, a impérativement besoin d’une amorce 1 (en vert). L’amorce est une courte séquence d’ADN complémentaire au début de la séquence que l’on veut voir copiée ❶. Cette amorce doit s’apparier exactement avec la séquence à copier : en face de A, l’amorce doit afficher un T, en face de T un A, en face de C un G et en face de G un C. Cet appariement est très solide et sa composition est dictée par la séquence de l’ADN viral. Il faut bien sûr connaître la séquence de l’ARN viral d’origine et choisir un endroit de cette séquence que le virus ne partage avec aucun autre virus. ❷ Avec l’aide d’une deuxième amorce, un fragment, de longueur et de séquence connues, peut être copié et amplifié par le nombre adéquat de cycles ❸.
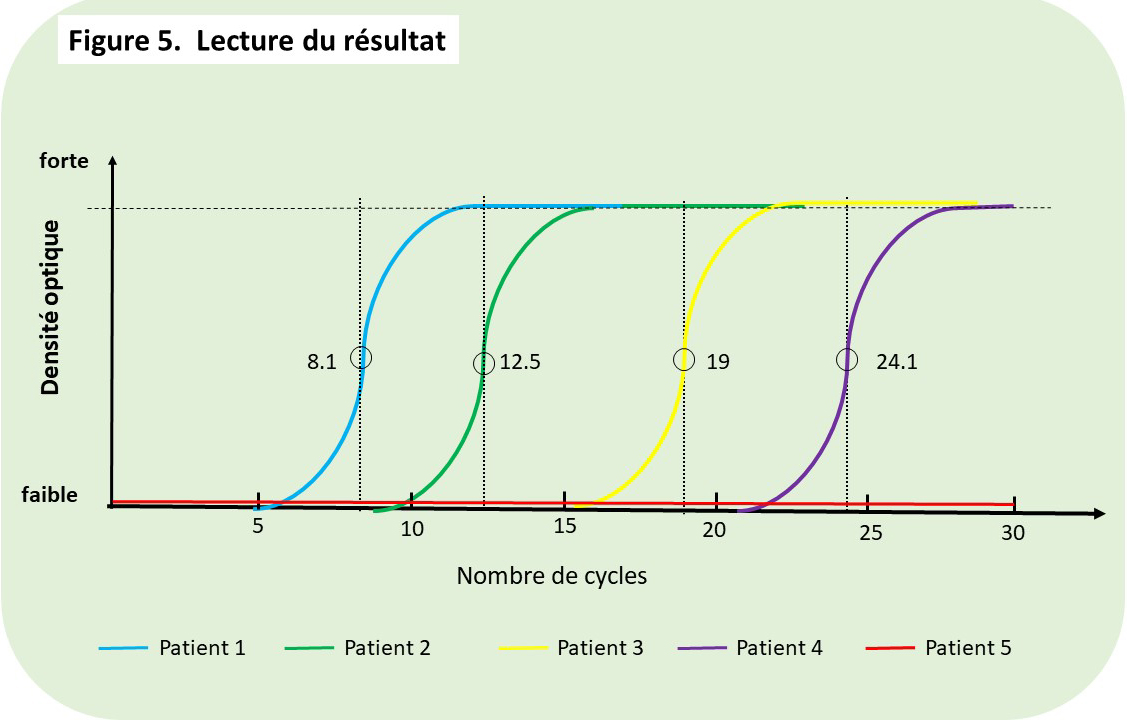
- La densité optique (coloration) augmente selon une cinétique sigmoïdale et atteint un maximum correspondant à la limite du fonctionnement de l’ADN polymérase qui a épuisé toutes ses ressources (Figure 5, plateau supérieur de toutes les courbes). Le nombre de cycles effectués pour atteindre cette limite varie. Plus il y a d’ADN viral au départ, plus rapidement la limite est atteinte. On exprime en général le résultat comme le nombre de cycles nécessaires pour atteindre le 50% de la valeur maximale.
- Dans la Figure 5, pour le patient 1, ce 50% du maximum est atteint après 8.1 cycles, pour le patient 2 après 12.5 cycles etc. Le patient 5 est négatif (pas d’amplification). La mise au point du test doit définir également le nombre de cycles à partir duquel le test après amplification est considéré comme négatif. En effet un produit d’amplification obtenu après un nombre de cycles très élevé (30 cycles = amplification d’un milliard de fois ) a une probabilité très élevée d’être un produit non spécifique. Une analyse du produit d’une telle amplification montrerait toute une série de fragments avec des tailles et des séquences indéterminées.
Le résultat d’un test, pour de vrai, est présenté à la Figure 6. On y voit un nombre de cycles de 18.5 et une note (a) qui rend attentif à un nombre trop élevé de cycles .
La calibration d’un tel test avec des quantités connue d’ADN viral permet de conclure au nombre de génomes viraux présent dans l’échantillon du patient (ce qui n’est pas fait en routine dans le test du SARS-CoV-2). Cette méthode est très utilisée pour suivre l’effet thérapeutique des médicaments antiviraux (HIV).
En conclusion, le test RT-PCR a une très grande spécificité grâce aux amorces qui, si elles sont judicieusement choisies, peuvent distinguer entre des virus très proches. Grâce à ses capacités d’amplification du signal, il affiche également une très bonne sensibilité. Ce test souffre pourtant du défaut de ses qualités. Sa sensibilité extrême et la grande quantité du produit amplifié l’exposent au risque de contamination des échantillons par les produits amplifiés. Par exemple, le laboratoire de préparation des réactions de RT-PCR doit impérativement être séparé de celui où les réactions d’amplification sont réalisées. C’est au final un test très technique qui demande rigueur et professionnalisme