 En gros. La génétique appliquée part généralement d’un caractère particulier d’un organisme vivant pour remonter à l’identification d’un ou de plusieurs gènes potentiellement responsables de cette caractéristique. L’opération de génétique inverse consiste à exprimer/réprimer ou modifier un ou des gènes pour analyser ou confirmer son rôle dans le caractère particulier. La génétique inverse nécessite l’accès physique au matériel génétique de l’organisme, son génome. Si dans le cas des dinosaures, l’accès au génome ainsi que l’environnement dans lequel les gènes sont exprimés posent quelques problèmes, l’opération pour les virus est plus simple. Le nombre de gènes est restreint (moins d’une dizaine à une centaine), et l’infection de cellules en cultures est suffisante pour les exprimer et, souvent, estimer leur rôle.
En gros. La génétique appliquée part généralement d’un caractère particulier d’un organisme vivant pour remonter à l’identification d’un ou de plusieurs gènes potentiellement responsables de cette caractéristique. L’opération de génétique inverse consiste à exprimer/réprimer ou modifier un ou des gènes pour analyser ou confirmer son rôle dans le caractère particulier. La génétique inverse nécessite l’accès physique au matériel génétique de l’organisme, son génome. Si dans le cas des dinosaures, l’accès au génome ainsi que l’environnement dans lequel les gènes sont exprimés posent quelques problèmes, l’opération pour les virus est plus simple. Le nombre de gènes est restreint (moins d’une dizaine à une centaine), et l’infection de cellules en cultures est suffisante pour les exprimer et, souvent, estimer leur rôle.
La génétique inverse est ainsi une méthode puissante pour identifier sans faille le gène responsable d’un caractère dit monogénique (qui ne dépend que d’un seul gène). Elle s’applique également pour les traits polygéniques, même si, dans ce cas, les opérations de vérification sont plus complexes. En plus de ces opérations de vérification, elle permet de modifier le génome d’un virus, lui conférant ainsi une nouvelle propriété. Cette nouvelle propriété peut advenir d’un retrait (délétion), d’un ajout (insertion) ou d’une modification (mutation) d’un gène.
L’ADN est le support physique de l’information génétique de tous les organismes vivants (cf. Pré-Requis). L’ARN intervient dans ce cadre comme le moyen de transférer l’information contenue dans l’ADN à la machinerie qui synthétise les protéines. On parle d’ARN messager, ARNm. Dans le cas des virus, s’il existe des virus dont le génome est fait d’ADN (virus à ADN), il existe également des virus dont le génome est composé d’ARN (virus à ARN). Pour ces derniers, on en distingue différentes catégories. L’ARN peut directement contenir l’information nécessaire à la synthèse des protéines, on parle de virus à ARN de sens positif {ARN(+)}. L’ARN peut être complémentaire à l’ARN contenant l’information, ce sont des virus à ARN de sens négatif {ARN(-)}. Une troisième catégorie concerne des virus à ARN double brin dont le génome est composé des deux brins d’ARN complémentaires {ARN(+)/ARN(-)}. Pour la dernière catégorie, celle des rétrovirus, le génome est composé d’ARN de sens positif, mais cet ARN (+) ne sert de messager qu’après avoir été converti en ADN duquel il est finalement produit.
L’ADN peut être « bricolé”. Dès les années 1950, la biologie moléculaire a développé des outils qui permettent de travailler l’ADN. Deux opérations essentielles deviennent possibles : la lecture de la séquence des quatre éléments qui le constituent (A,T,G,C) et l’identification de ciseaux (enzymes de restriction) qui coupent l’ADN à des endroits très précis formés par une séquence de quelques lettres. Par exemple, l’enzyme de restriction BamH1 coupe à GGATCC et l’enzyme Xho1 à CTCGAG etc. La multiplication de ces enzymes a permis ainsi de fractionner l’ADN en petits morceaux qui pouvaient ensuite être recollés pour générer un ADN auquel un morceau avait été retiré ou ajouté, une opération que l’on a appelé un clonage. Durant les dernières décennies du XXème siècle, l’habileté à inventer des stratégies de clonage a représenté une qualité hautement appréciée. De nos jours, ce type de clonage a quasiment disparu car des synthétiseurs d’ADN mettent à disposition des chercheurs les molécules d’ADN contenant les séquences d’information voulue. De plus, des ciseaux hautement performants pouvant être dirigés vers des séquences d’ADN ciblées (Crispr-Cas9), ouvrent des possibilités de modification illimitées, non seulement au niveau des génomes viraux mais également des génomes d’organismes supérieurs, y compris l’humain.
L’ARN n’est pas “bricolable« . L’ARN est une molécule extrêmement fragile, pour laquelle l’évolution n’a pas sélectionné d’outils pour la modifier. De plus, ce n’est que très récemment (cf. réf de 2025*) qu’une méthode de séquence directe d’ARN a été proposée. L’évolution n’a clairement pas envisagé qu’un jour des chercheurs aimeraient aussi bricoler les génomes viraux à ARN. Pour le faire, le génome à ARN doit, en laboratoire, être copié en ADN. Ceci est possible grâce à une enzyme purifiée des cellules infectées par un rétrovirus (rétro-transcriptase). Une fois sous cette forme, les outils pour le transformer sont ceux qui s’appliquent à l’ADN. Reste au final, la production de l’ARN modifié à partir de l’ADN modifié. Et cela se fait grâce à des outils biologiques existants, des ARN polymérases dépendantes de l’ADN. Ces enzymes, semblables à celles produisant les ARNm, peuvent simplement être achetées sur le marché des enzymes.
Côté pratique : génétique inverse pour virus à ADN. Partant d’une préparation virale, généralement obtenue par culture du virus en laboratoire, quoiqu’un échantillon venant d’un organisme infecté puisse suffire, la séquence de l’information génétique est obtenue grâce à un « séquenceur », un appareil conçu pour lire la suite des 4 lettres qui la constitue. Une fois la séquence d’information connue, le synthétiseur d’ADN peut être programmé et un génome fait d’ADN peut être produit. A cette étape, des modifications peuvent être faites (cf. Figure 1). Ce génome est ensuite introduit dans les cellules en culture, une opération nommée « transfection », où l’ADN est enrobé dans une coque de graisse qui permet de franchir les barrières cellulaires. L’ADN ainsi introduit doit atteindre le noyau où il va être transcrit en ARN messagers viraux, comme lors d’une infection, et la production de virus modifiés est enclenchée.
Ainsi décrite, l’opération semble toute simple. Pourtant, ce n’est pas forcément le cas. Lors d’une infection virale, le virus entre dans la cellule avec son génome certes, mais avec d’autres protéines virales qui vont maximiser l’opération. Exemple : un ADN qui se ballade hors du noyau de la cellule est un signal d’effroi qui enclenche une réaction cellulaire de défense qui passe ultimement par un suicide cellulaire. Dans une infection naturelle, le génome viral est accompagné, caché, par des protéines virales jusqu’à être injecté dans le noyau cellulaire, sans avoir été détecté hors du noyau. De fait, la génétique inverse pour des virus à ADN n’est limitée que par la nécessité d’envoyer l’ADN modifié en laboratoire dans le noyau. L’opération n’est pas forcément très efficace, mais elle repose sur la puissance d’amplification d’une seule réussite sur plusieurs millions d’échecs. Il suffit d’une cellule où la production virale est réussie pour que les milliers de virus produits infectent à terme la culture et produisent les milliards de virus attendus.
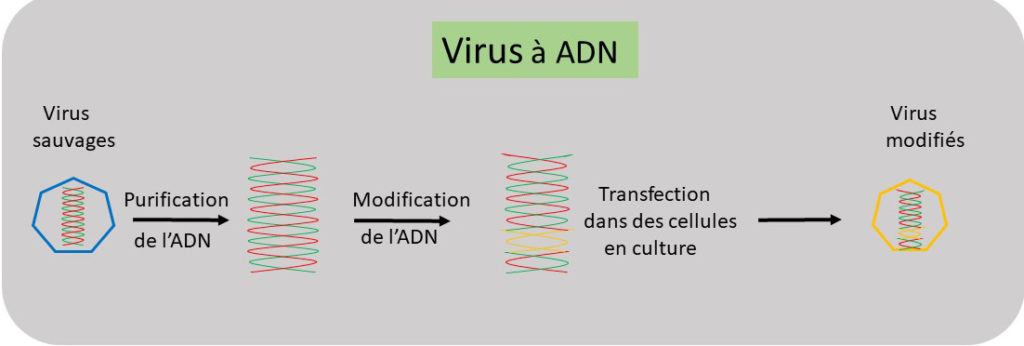
Figure 1. L’ADN viral est purifié et sa séquence obtenue. L’ADN est ensuite produit avec les modifications voulues. L’ADN modifié est introduit dans les cellules pour générer des virus portant les modifications voulues.
Côté pratique : génétique inverse pour les virus à ARN. Comme indiqué ci-dessus, les virus à ARN viennent en différentes catégories. Commençons par la catégorie la plus simple, celle des virus à ARN de sens positif {ARN(+)}. Ici le génome viral représente l’ARNm. Il suffit donc pour produire un virus d’introduire (transfection) dans les cellules un exemplaire de ce génome pour que le virus soit produit. Sauf que, l’idée ici est de pouvoir modifier ce génome pour produire une version modifiée du virus. Pour cela, il faut produire une version ADN du génome. Pour ce faire, une enzyme virale provenant d’un rétrovirus (rétro-transcriptase), et disponible sur le marché, effectue une rétro-transcription, c’est à dire une copie ADN du génome ARN. La version ADN peut maintenant être modifiée à l’envi. De cette version ADN modifiée, une version ARN modifiée est produite par transcription cette fois (une opération faite par une enzyme disponible sur le marché, la T7 polymérase) et est introduite par transfection dans les cellules, actionnant la production du virus modifié. Dans les faits, plutôt qu’introduire l’ARN viral par transfection, c’est la version ADN qui est introduite dans des cellules elles-mêmes modifiées pour exprimer la T7 polymérase. La production de ce type de cellules a été une avancée majeure dans le champ de la génétique inverse des virus à ARN. C’est le chercheur allemand Karl-Klaus Conzelmann qui a réussi cet exploit. Il a mis gratuitement à disposition de la communauté virologique la lignée cellulaire BSR T7, exprimant la polymérase T7.
Pour les virus à ARN de sens négatif {ARN(-)}, le génome est le complément des ARNm. De plus, ce génome, est étroitement associé à une des protéines virales (NP), sous forme de nucléocapside, une nucléocapside qui constitue le génome actif du virus. L’ARN viral est ici produit, à partir d’une version ADN comme ci-dessus, dans le contexte des nombreuses NP, dont il va falloir également introduire le gène. Finalement, lorsque l’ARN viral est entouré de NP, l’enzyme virale qui lit la nucléocapside (une ARN polymérase virale) pour produire les mARN doit également être introduite. C’est donc une transfection à trois éléments qui est nécessaire pour enclencher la production du virus.
Les virus dont le génome est composé d’ARN double brin (dbARN), ont la particularité de voir ce génome composé de plusieurs segments (10 ou plus). Au final, une transfection de fragments d’ADN contenant une version fidèle de chaque segment et de son complément ont permis d’obtenir avec efficacité des virus. Ici, il s’agit donc d’une opération à deux dizaines (ou plus) d’éléments.
Pour les rétrovirus, dont le génome est bien une molécule d’ARN de sens positif, le processus naturel d’infection commence par la production à partir de cette molécule d’ARN d’une version ADN double brin de ce génome (provirus). Le provirus se voit obligatoirement intégré dans le génome de la cellule infectée. De cette forme intégrée, les ARNm du virus ainsi que le génome ARN sont produits. L’opération de génétique inverse consiste dans ce cas en l’introduction d’une version ADN du génome viral, sous une forme qui va permettre aux fonctions cellulaires de produire les ARNm du virus et l’ARN génomique qui sera intégré dans les particules virales.
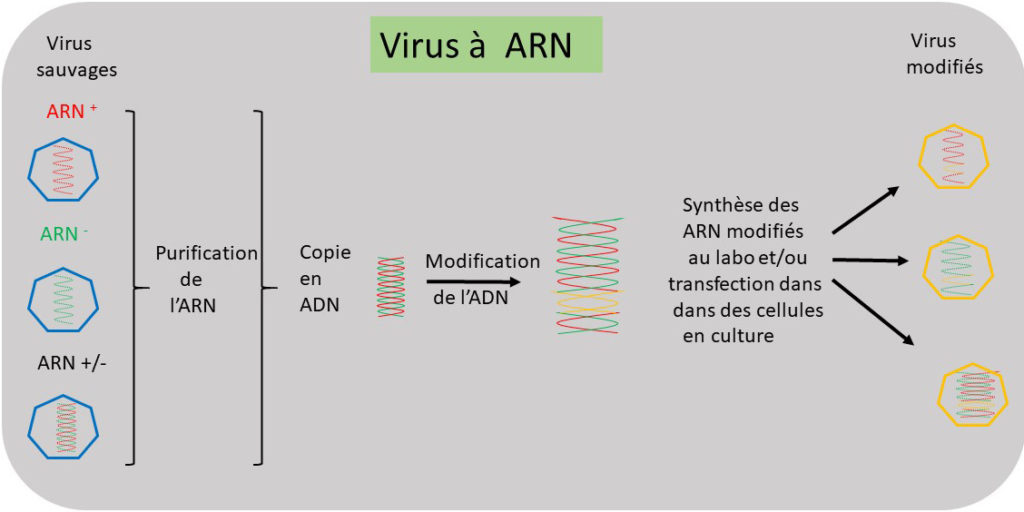
Figure 2. La modification d’un génome à ARN, passe par une version ADN de ce génome, qui lui peut subir les modifications. Ces génomes ADN modifiés peuvent ensuite servir à la synthèse de leurs versions ARN, qui introduites dans les cellules vont permettre de produire les virus modifiés (cf. texte).
En guise de conclusion. Il est ainsi possible de nos jours d’appliquer les techniques de génétique inverse à la majorité des virus connus à ce jour. Comme indiqué en introduction, ces techniques permettent d’identifier de manière très efficace les fonctions des protéines virales, en observant l’effet de leur suppression ou de leur modification. Elles permettent la production de virus avec des propriétés nouvelles. Il n’est que de citer la production de virus dont la virulence est atténuée, utilisés en guise de vaccin. Vaccins contre le virus homologue sauvage ou vaccin contre un autre virus dont une ou des protéines auraient été intégrées et présentées par le virus atténué. Le virus restant un champion de l’introduction de gènes dans les cellules, il peut être utilisé en thérapie génique en introduisant efficacement le gène déficient ou manquant qu’il faut remplacer. Le virus restant un destructeur efficace des cellules infectées, il peut être ciblé par adjonction d’une protéine exprimée par des cellules cancéreuses et devenir un tueur efficace des cellules ciblées, on parle ici de virus oncolytiques. Ainsi vaccinologie, thérapie génique et destruction ciblée de cellules cancéreuses sont des domaine ouverts à la réalisation de solutions par la génétique inverse des virus.
*Sarah E. Pye et al. J Virol. 2025 Aug 19;99(8):e0089425. doi: 10.1128/jvi.00894-25.
Publié le 29 juillet 2025, màj. le 17 janvier 2026.